典型相关分析介绍及python实现
在处理单个高维数据时,通过可以通过LDA,PCA,等等方法进行降维处理,但是如果某两个数据来自同一个样本,但是数据类型不同,差距巨大时,怎么办呢?这个时候就是典型相关性分析(Canonical Correlation Analysis,CCA)的应用场景.CCA允许我们同时从两套数据分析.典型的应用场景就包括生物学上的联合分析,同一组样本,同时检测转录组和蛋白组,转录组和代谢组以及微生物代谢组等等,更详细的内容可参考维基百科.
CCA与PCA的联系与差别
CCA有点类似PCA(主成分分析,principal component analysis),它们都由同一个课题组提出,在降维方面(canonical variables)可以认为是多套数据的PCA.
不同之处是PCA旨在找出一套数据中能够表示最多方差的线性组合,而CCA旨在找出两套数据中能够最大程度表示其相关性的线性组合.
python实现CCA分析
那么在python中如何实现CCA分析呢? 在sklearn包中的cross_decomposition提供了CCA分析方法,直接调用即可,这里以企鹅数据为例
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
import numpy as np
filename = "penguins.csv"
df = pd.read_csv(filename)
df =df.dropna()
df.head()
展示数据
species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g sex
0 Adelie Torgersen 39.1 18.7 181.0 3750.0 MALE
1 Adelie Torgersen 39.5 17.4 186.0 3800.0 FEMALE
2 Adelie Torgersen 40.3 18.0 195.0 3250.0 FEMALE
4 Adelie Torgersen 36.7 19.3 193.0 3450.0 FEMALE
5 Adelie Torgersen 39.3 20.6 190.0 3650.0 MALE
我们选取其中两种特征作为研究对象bill_length_mm和bill_depth_mm作为一组,而flipper_length_mm与body_mass_g作为另一组,
from sklearn.preprocessing import StandardScaler
df1 = df[["bill_length_mm","bill_depth_mm"]]
df1_std = pd.DataFrame(StandardScaler().fit(df1).transform(df1), columns = df1.columns)
df1_std.head()
df2 = df[["flipper_length_mm","body_mass_g"]]
df2_std = pd.DataFrame(StandardScaler().fit(df2).transform(df2), columns = df2.columns)
df2_std.head()
数据变成
# df 1
bill_length_mm bill_depth_mm
0 -0.896042 0.780732
1 -0.822788 0.119584
2 -0.676280 0.424729
3 -1.335566 1.085877
4 -0.859415 1.747026
# df2
flipper_length_mm body_mass_g
0 -1.426752 -0.568475
1 -1.069474 -0.506286
2 -0.426373 -1.190361
3 -0.569284 -0.941606
4 -0.783651 -0.692852
开始正式的CCA分析
from sklearn.cross_decomposition import CCA
ca = CCA()
xc,yc = ca.fit(df1, df2).transform(df1, df2)
其得到的结果与输入长度一致
np.shape(xc)
# (333,2)
np.shape(yc)
# (333,2)
而且是高度相关的,这里直接计算其相关性
np.corrcoef(xc[:, 0], yc[:, 0])
np.corrcoef(xc[:, 1], yc[:, 1])
相关性结果为
array([[1. , 0.78763151],
[0.78763151, 1. ]])
array([[1. , 0.08638695],
[0.08638695, 1. ]])
第一对结果很好,相关性0.79,而第二对结果堪忧,我们将CCA分析得到的结果与物种和性别组合成新的dataframe
cca_res = pd.DataFrame({"CCA1_1":xc[:, 0],
"CCA2_1":yc[:, 0],
"CCA1_2":xc[:, 1],
"CCA2_2":yc[:, 1],
"Species":df.species,
"sex":df.sex})
cca_res.head()
其数据为
CCA1_1 CCA2_1 CCA1_2 CCA2_2 Species sex
0 -1.186252 -1.408795 -0.010367 0.682866 Adelie MALE
1 -0.709573 -1.053857 -0.456036 0.429879 Adelie FEMALE
2 -0.790732 -0.393550 -0.130809 -0.839620 Adelie FEMALE
4 -1.718663 -0.542888 -0.073623 -0.458571 Adelie FEMALE
5 -1.772295 -0.763548 0.736248 -0.014204 Adelie MALE
通过其第一列数据进行散点图分析
sns.scatterplot(data=cca_res,x="CCA1_1",y="CCA2_1",hue="Species",s=10)
plt.title(f'First column corr = {np.corrcoef(cca_res.CCA1_1,cca_res.CCA2_1)[0, 1]:.2f}')
plt.savefig("cca_first.png",dpi=200)
plt.close()
其图为:
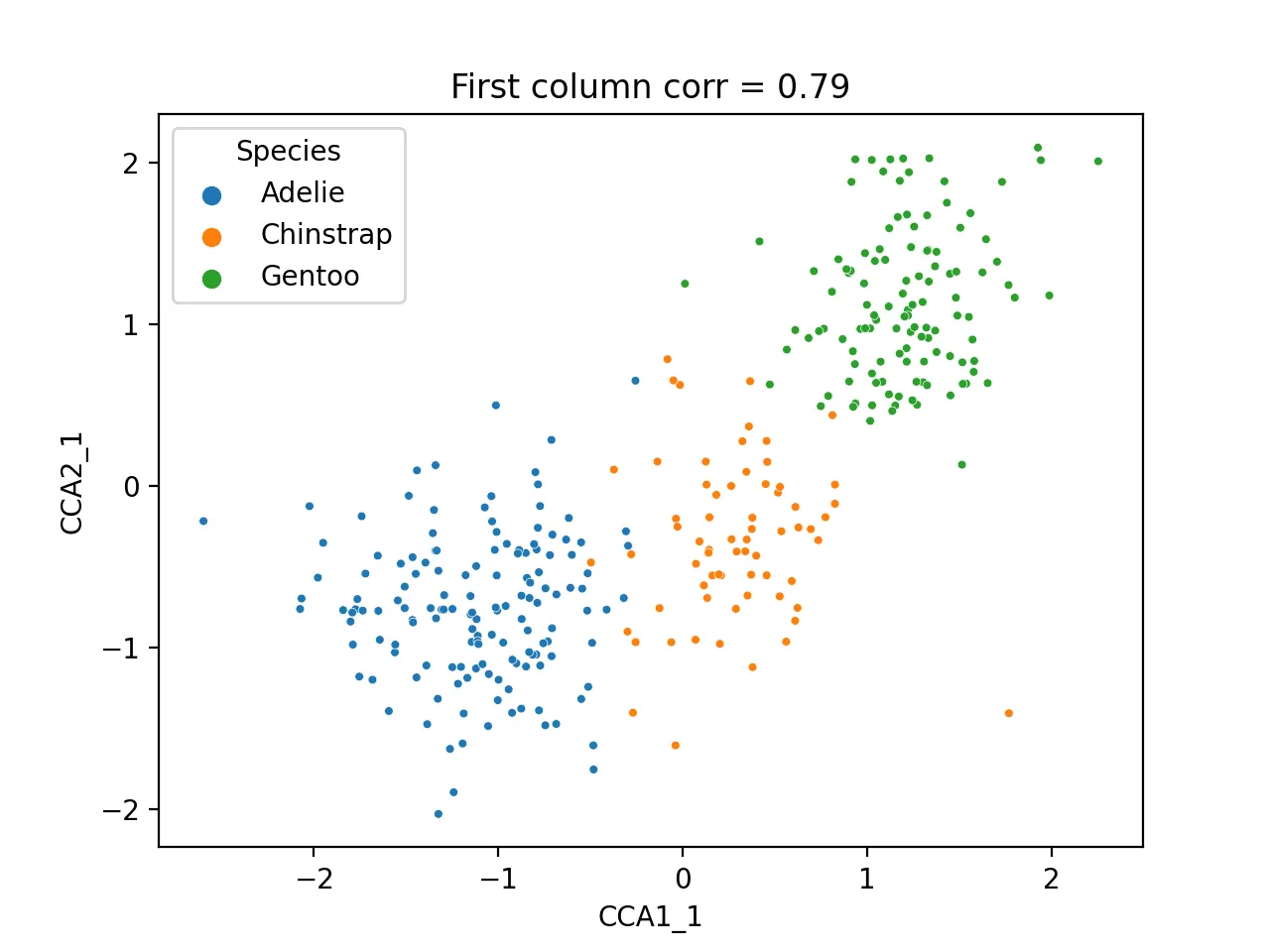 鉴于第二列数据相关性低,这里通过与原始矩阵进行相关性热图比较,找到其可能的体现的数据
鉴于第二列数据相关性低,这里通过与原始矩阵进行相关性热图比较,找到其可能的体现的数据
cca_df = pd.DataFrame({"cca1_1":cca_res.CCA1_1,
"cca1_2":cca_res.CCA1_2,
"cca2_1":cca_res.CCA2_1,
"cca2_2":cca_res.CCA2_2,
"Species":df.species.astype('category').cat.codes,
"Island":df.island.astype('category').cat.codes,
"sex":df.sex.astype('category').cat.codes})
dfcor = cca_df.corr()
mask = np.triu(np.ones_like(dfcor))
sns.heatmap(dfcor,cmap="bwr",annot=True)
plt.savefig("cca_corr.png",dpi=200)
plt.close()
绘制的图为:
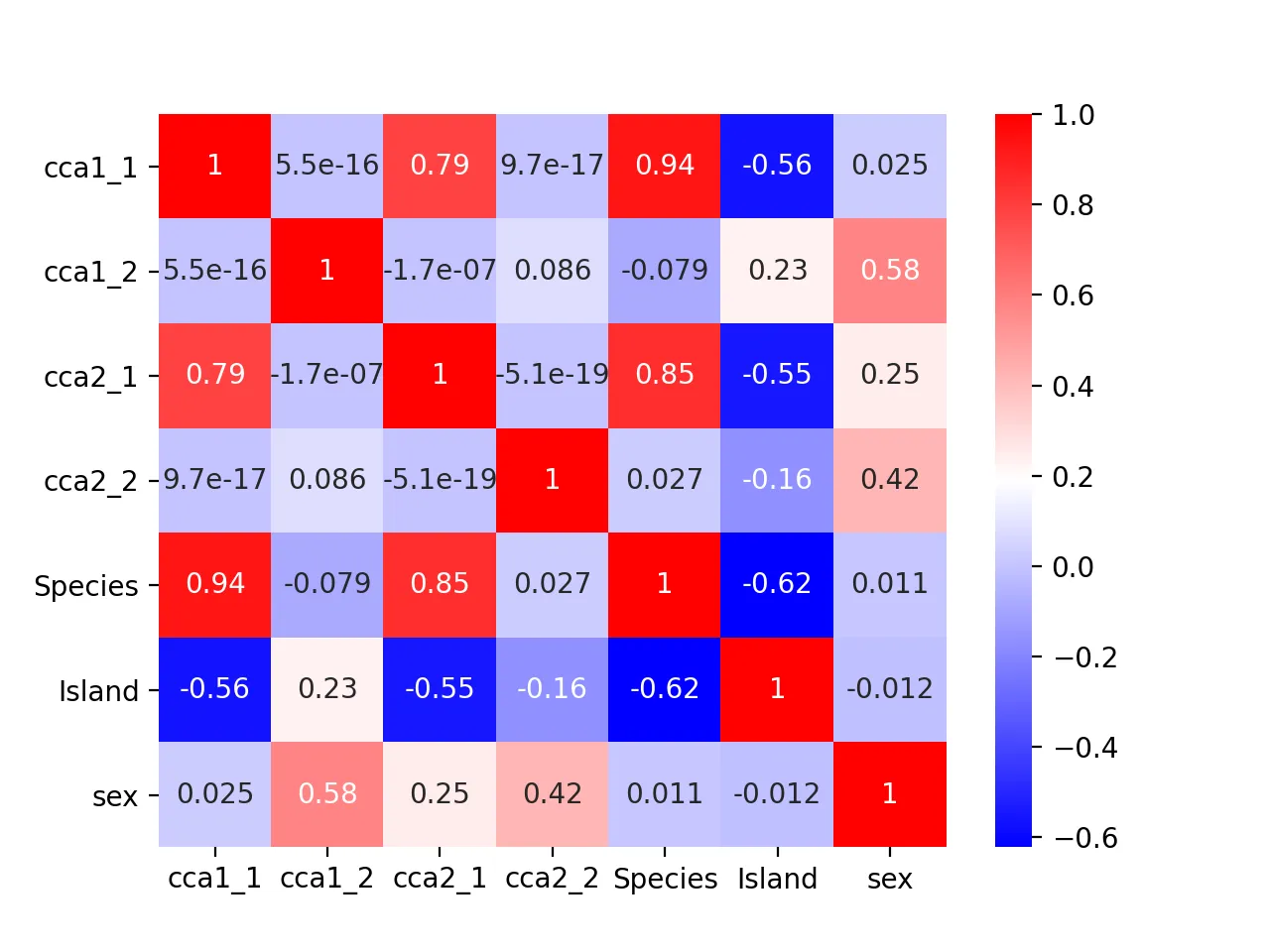
可以看到sex性别与cca2_2相关性最大,为0.42,推测其隐藏信息是性别
sns.scatterplot(data=cca_res,x="CCA1_2",y="CCA2_2",hue="sex",s=10)
plt.title(f'Second column corr = {np.corrcoef(cca_res.CCA1_2,cca_res.CCA2_2)[0, 1]:.2f}')
plt.savefig("cca_sex.png",dpi=200)
plt.close()
图为:
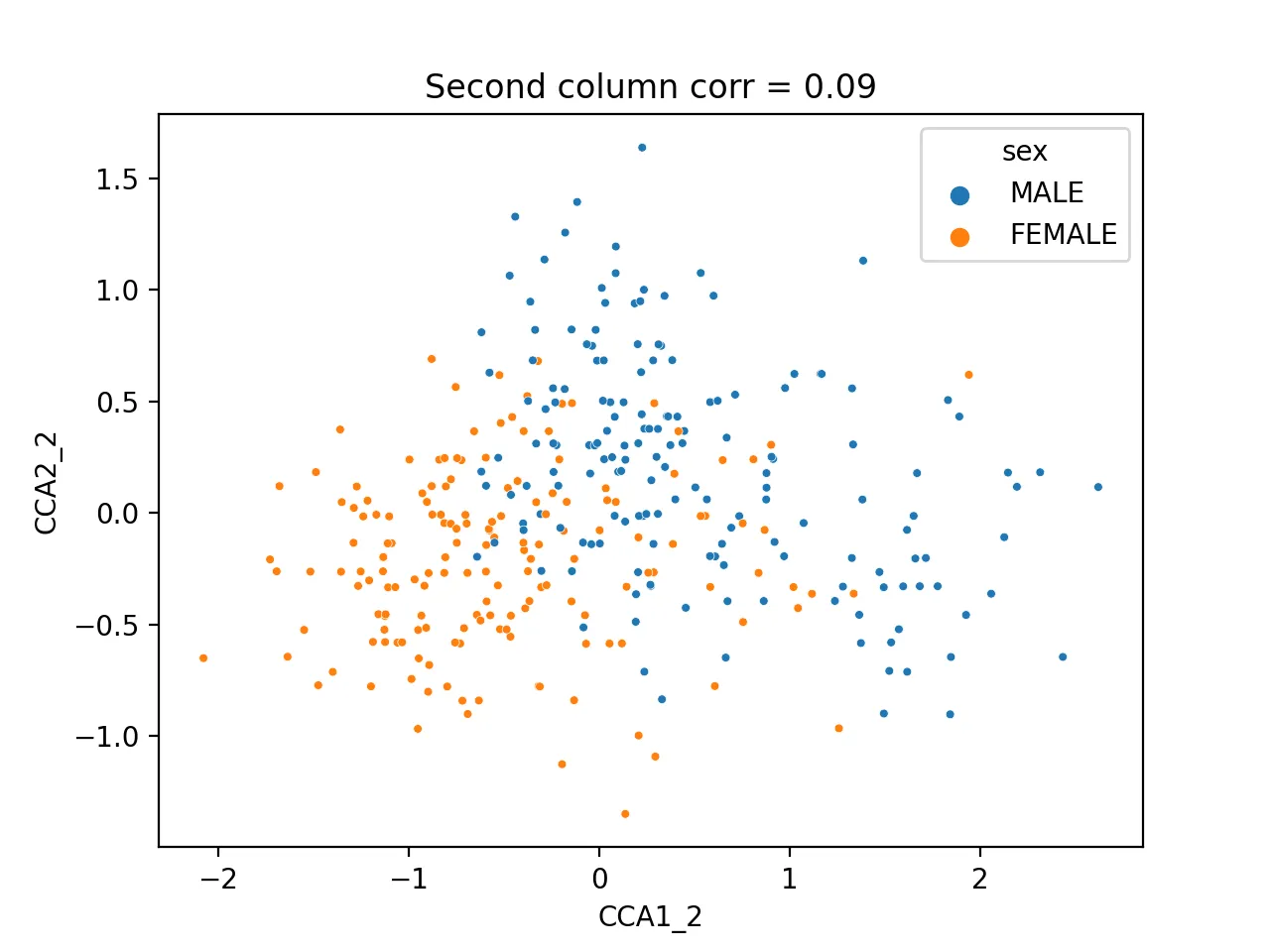 不同样本可以明显区分开来,
不同样本可以明显区分开来,
总结
CCA在高维数据中可以很好的进行多组类型数据的处理,在企鹅数据中表现很好,在生物学上多组学数据的联合分析也是基础的处理方法,后续继续介绍其他的多组学处理方法.
- 原文作者:春江暮客
- 原文链接:https://www.bobobk.com/581.html
- 版权声明:本作品采用知识共享署名-非商业性使用-禁止演绎 4.0 国际许可协议进行许可,非商业转载请注明出处(作者,原文链接),商业转载请联系作者获得授权。